+49 (0) 3727 / 58-1242
Lasertechnologien
Biophotonik
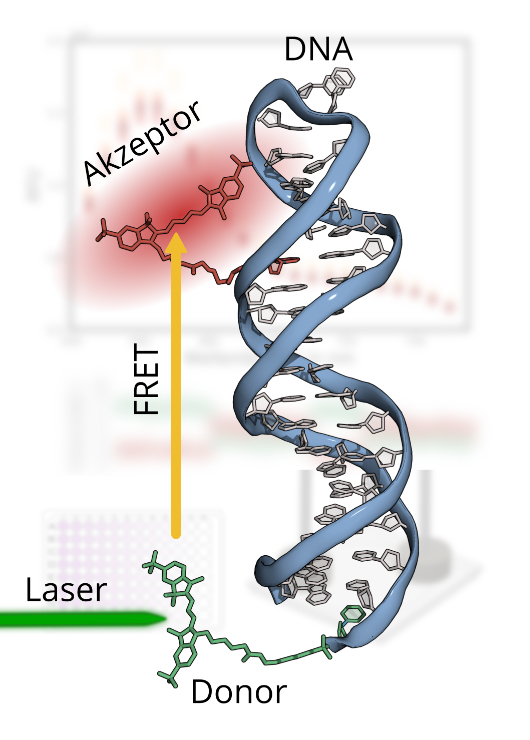
Die Biophotonik beschäftigt sich allgemein mit der Wechselwirkung von Licht mit organischer Materie. Am LHM liegt der Fokus speziell auf der Interaktion von Licht mit einzelnen Biomolekülen. Dabei wird sichtbare Laserstrahlung genutzt, um farbstoffmarkierte Biomoleküle, insbesondere RNA und deren Interaktionspartner zu untersuchen. Am LHM wird nicht nur eine vollständige Produktionsstrecke entwickelt, um das zu untersuchende RNA-Biomolekül herzustellen und fluoreszent zu markieren, sondern auch eine vollständig automatisierte Probenpräparation für die Fluoreszenzmikroskopie/-spektroskopie. Der Messwert der Wahl ist der Fluoreszenz-Resonanz-Energietransfer, kurz FRET, ein Distanzmaßstab auf Molekülebene. Das FRET-Signal liefert sowohl Distanz als auch Dynamik/Kinetik-Informationen des untersuchten Biomoleküls. Diese Information werden genutzt, um dynamische 3D-Strukturmodelle herzustellen und diese in Abhängigkeit von der Umgebung und der Interaktionspartner zu untersuchen. Letzteres gelingt durch den Einsatz von Molekulardynamik-Simulationen und de novo Modellierung, die am LHM aus Anwendersicht betrieben und weiterentwickelt wird. Diese Forschung soll der pharmazeutischen Industrie in der Entwicklung neuartiger Medikamente und der medizinischen Diagnostik von RNA-basierten Diagnoseverfahren im Transferprozess zugänglich gemacht werden.
RNA-Herstellung und Fluoreszenzmarkierung
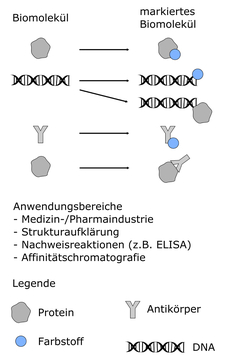
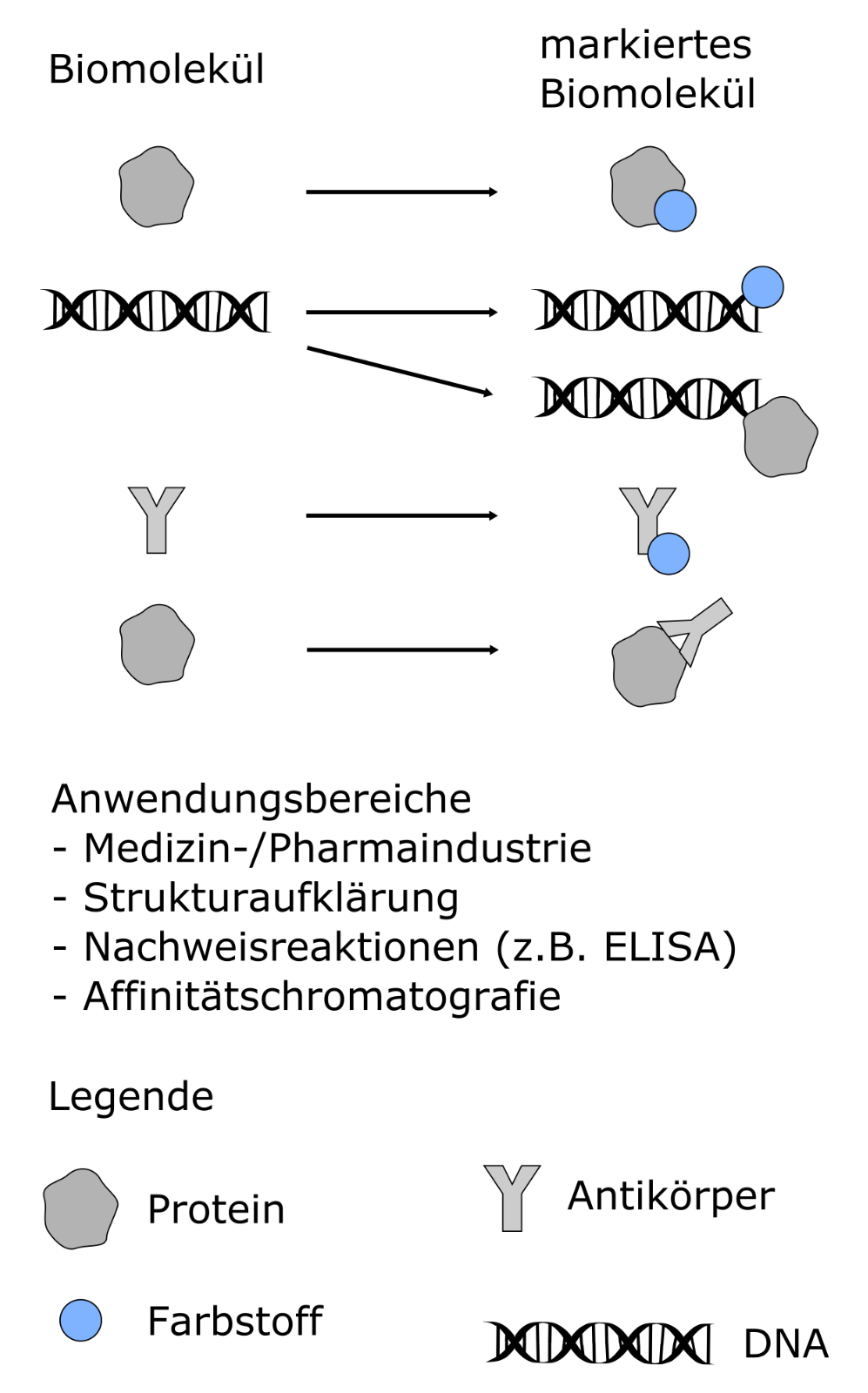
Unter der Markierung von Biomolekülen versteht man das Anfügen von Atomen, chemischen Gruppen oder Molekülen an Biomoleküle wie z.B. Proteine, DNA oder RNA. Dabei ist grundsätzlich zu unterscheiden, an welchem Molekül und an welcher Position das Label angebracht werden soll. So gibt es z.B. unterschiedliche Methoden für das Markieren von Proteinen oder das Markieren von Nukleinsäuren. Ebenso sind die Techniken unterschiedlich, ob die Markierung am Anfang/Ende der Molekülsequenz oder innerhalb der Sequenz erfolgen soll. Das Markieren von Biomolekülen dient oft zur Strukturaufklärung oder zur Verfolgung des markierten Moleküls für die Untersuchung von Reaktionsmechanismen und -kinetiken. Am LHM wird an der enzymatischen 3'-Ende/5'-Ende-Markierung von RNA-Molekülen mit fluoreszierenden Farbstoffen, z.B. Carbocyaninen, gearbeitet. Die farbstoffmarkierten RNA-Moleküle werden mittels FRET untersucht. Ziel ist es, durch die Bewegungs- und Distanzinformation der Farbstoffe unter verschiedenen Umgebungsbedingungen, Aussagen über deren Zustand treffen zu können. Dazu wird eine kostengünstige und effiziente Methode benötigt, um die Farbstoffe an das RNA-Molekül anzubringen.
Mikrofluidik für die automatisierte Probenpräparation in der Einzelmolekülfluoreszenzmikroskopie
Die richtige Probenpräparation ist ein wesentlicher Erfolgsgarant für die Einzelmolekülfluoreszenzmikroskopie und umfasst i.d.R. mehrere Schritte, die oft von erfahrenen Laborant:innen ausgeführt werden müssen. Dabei ist die Passivierung der Probenkammeroberfläche und die anschließende Immobilisierung der Einzelmoleküle ein komplexes Verfahren, bei dem verschiedene Puffer- und Präparationslösungen in einer definierten Reihenfolge in die Probenkammer injiziert werden. Die manuelle Probenpräparation ist dabei anfällig für Fluoreszenzkontaminationen und Fehler bei der Durchführung z.B. der Reihenfolge der Applikation. Um die Qualität und Reproduzierbarkeit der Einzelmolekülpräparation zu verbessern, wird am LHM eine Druckluft-gesteuerte Mikrofluidik genutzt und die Probenpräparation vollständig automatisiert. Die Mikrofluidik baut auf kommerziell erhältlichen Probenkammern (Ibidi, Grace Bio-labs) und einer Software-gesteuerten Druckluftsteuerung (Elfeflow) auf, die entsprechend an die Bedürfnisse der Einzelmolekülspektroskopie angepasst wurde.
Neben dem Aufbau und der Software-Steuerung, werden verschiedene Materialien und Verfahren der additiven Fertigung (FDM, SLA, Mikro-SLM) für die Herstellung von Reservoir-Adaptern getestet.
Fluiddynamiksimulation
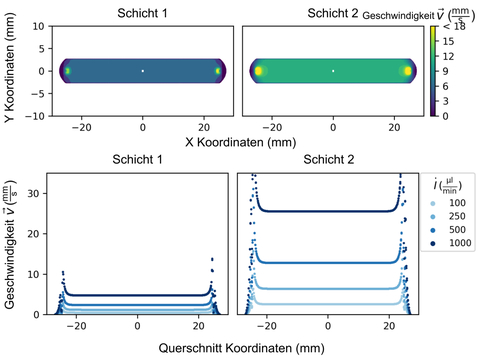
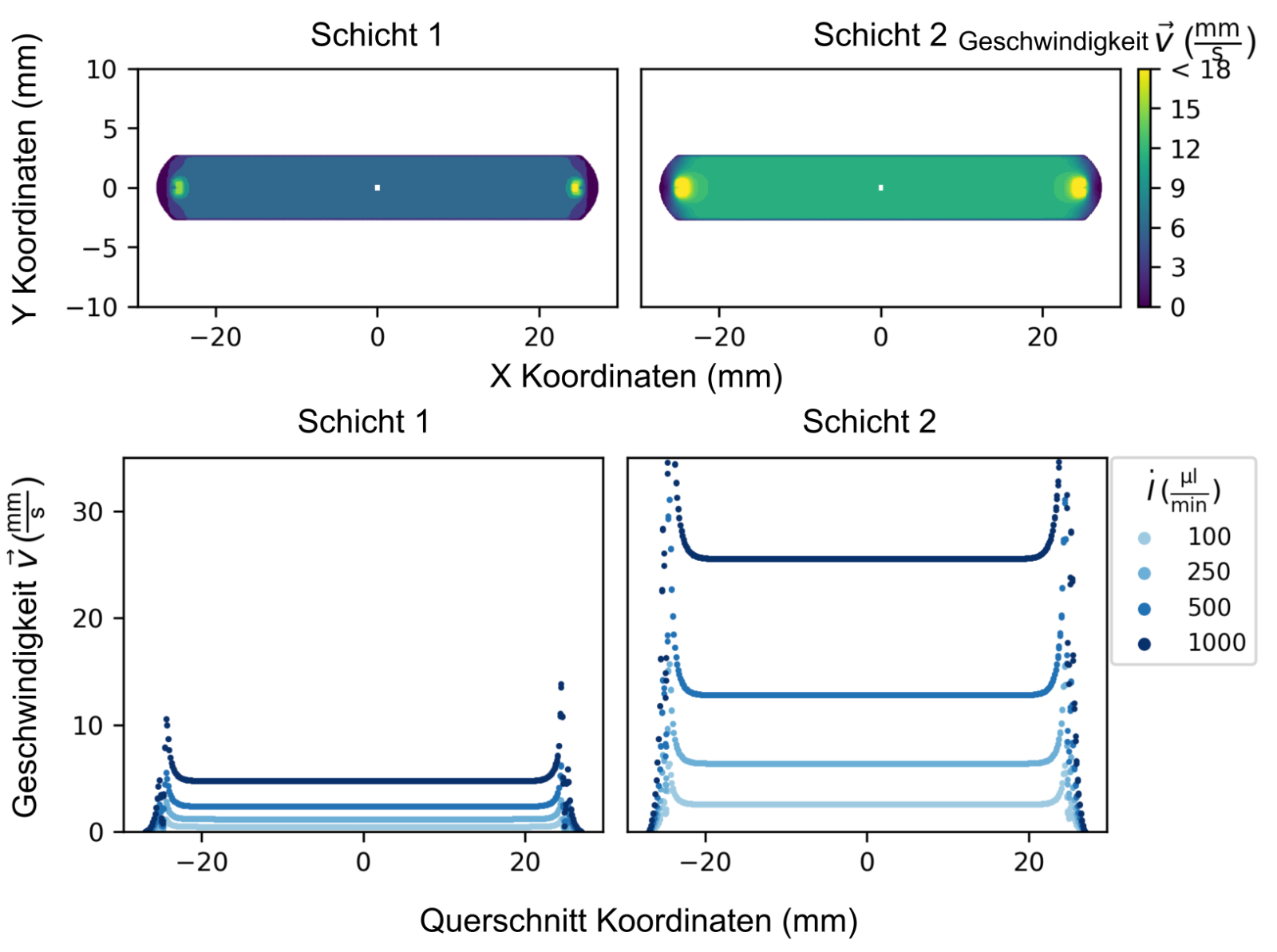
Flusssimulationen bieten die Möglichkeit, das Strömungsverhalten, sowohl von äußeren Konturen als auch durchströmten Kanälen, zu simulieren und zu bewerten. Durch die (Echtzeit-) Visualisierung unterstützt die Simulation sowohl den Arbeitsablauf als auch das Verständnis des Mikrofluidiksystems. Dadurch können Prozesse im Labor besser automatisiert und die Experimente optimiert werden.
Data Science
Am LHM werden in Forschungsprojekten große Mengen an Daten generiert. Um aus gewonnen Daten Informationen zu ziehen, ist es wichtig diese Daten zu visualisieren und effizient auszuwerten. Hierbei bietet die bioinformatische Auswertung der Daten mittels der Programmiersprache Python eine leistungsstarke Möglichkeit, komplexe und große Datenmengen zu verstehen und zu präsentieren. Die Labor-Ergebnisse können analysiert, interpretiert, weiterverarbeitet und übersichtlich dargestellt werden, da händische Berechnungen und Visualisierungen bei einer großen Datenmenge schnell an ihre Grenzen stoßen.
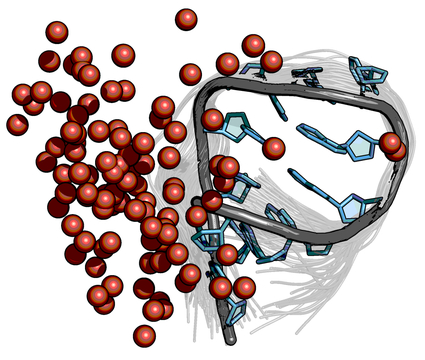
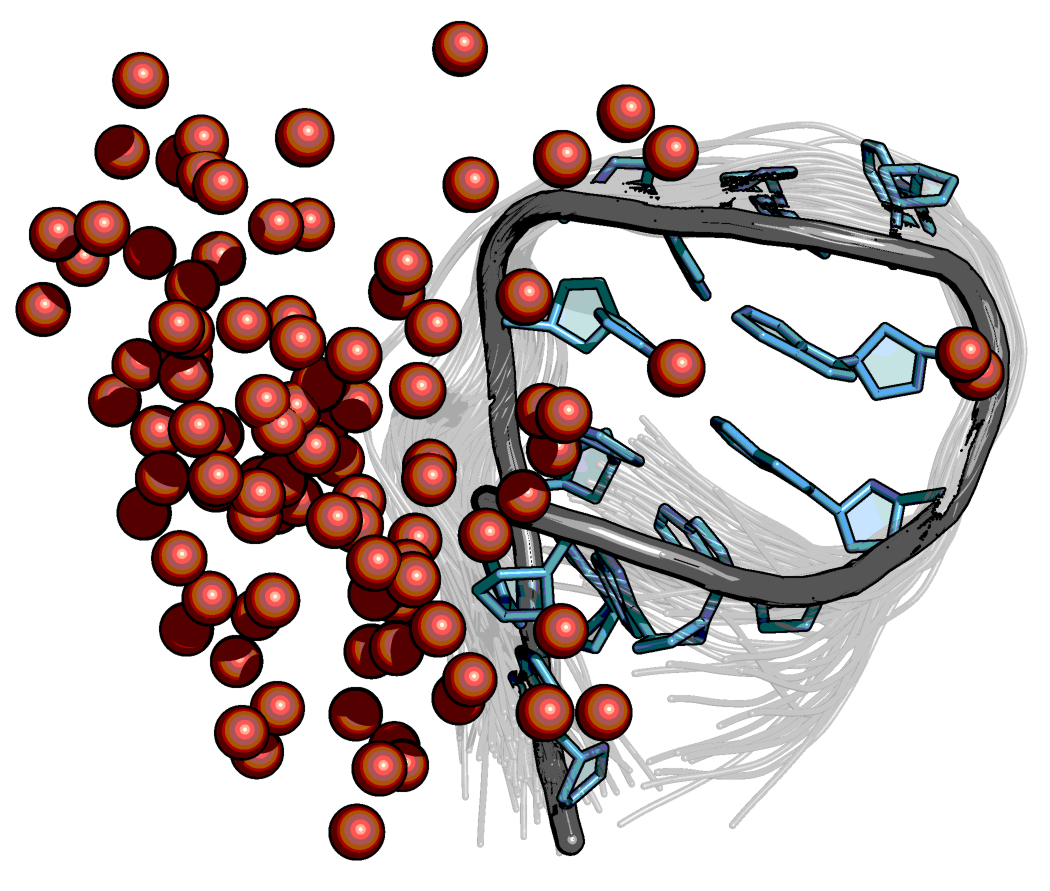
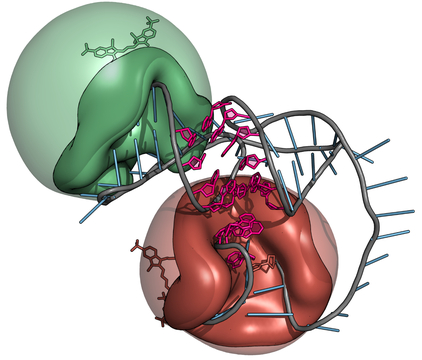
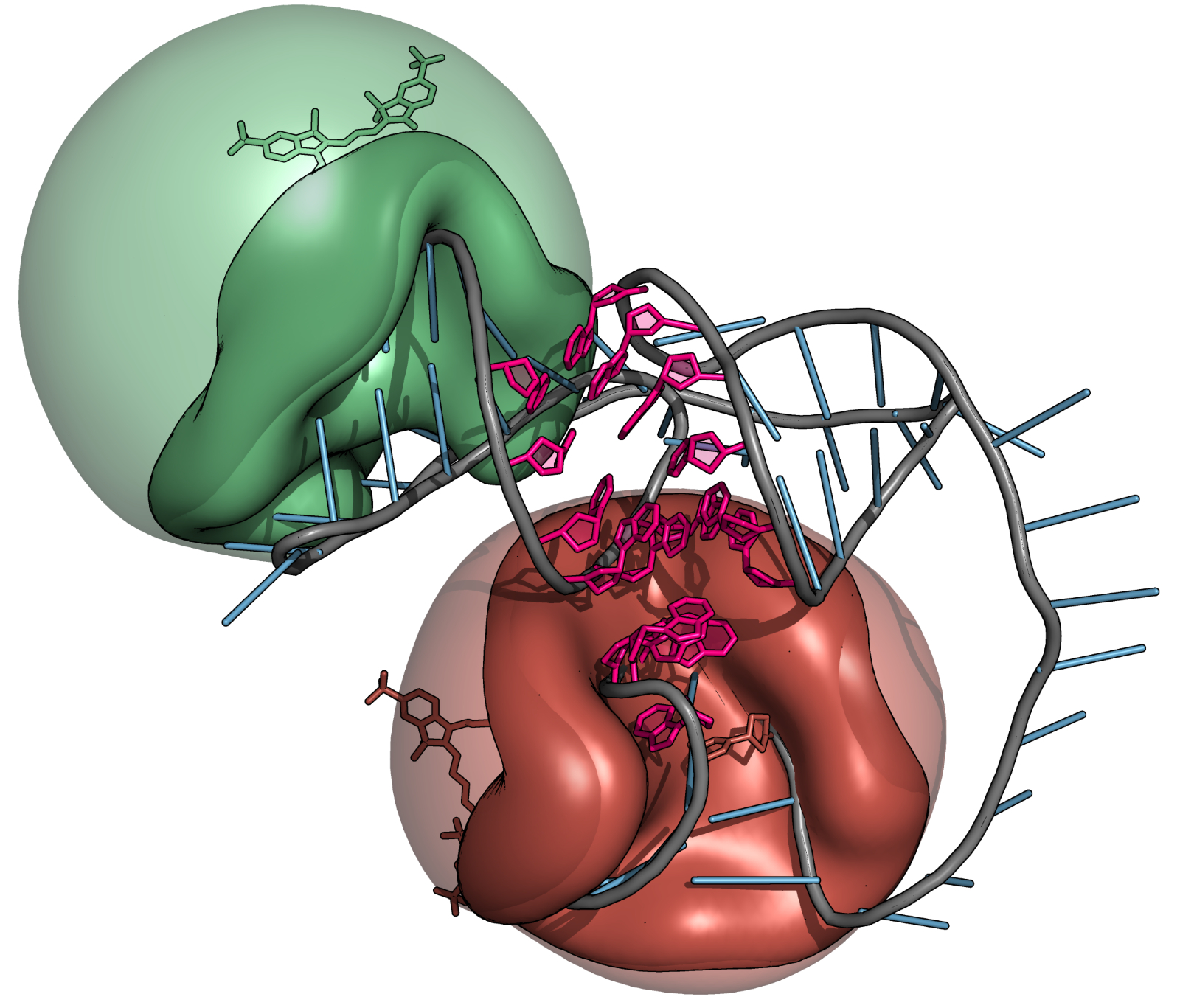
Integrative Strukturmodellierung und Simulation von RNA
Mithilfe von Computersimulationen können Experimente effizient geplant, interpretiert und sogar ganze Forschungsfelder bearbeitet werden. Mithilfe von integrativer Modellierung ist es möglich aus experimentellen Daten die Struktur von Biomolekülen vorherzusagen. Das Augenmerk am LHM liegt hierbei auf der Struktur- und Dynamikaufklärung von Ribonukleinsäuren (RNA) mithilfe des Förster Resonanz Energie Transfers (FRET). Ribonukleinsäuren übernehmen wichtige Funktionen im menschlichen Körper und sind daher interessant für die Medikamentenentwicklung. Mithilfe der am LHM entwickelten integrativen Modellierungpipeline soll die Suche nach potenziellen Medikamenten vereinfacht werden. Neben der Entwicklung neuartiger Methoden steht auch die Optimierung labortechnischer Verfahren im Fokus. Beispielsweise können mithilfe von RNA-Computermodellen mögliche Farbstoffmarkierungstellen vorhergesagt und evaluiert werden.
Die Struktur von Ribonukleinsäuren kann dazu beitragen wichtige Prozesse in Zellen besser zu verstehen. Durch integrative Strukturmodellierung mithilfe von experimentellen FRET-Daten können Strukturen der RNA aufgeklärt werden. Dabei werden de novo Modelle der RNA erstellt und die RNA-Bewegung und Farbstoffdynamik simuliert um anschließend die Strukturmodelle mithilfe von Experimentellen Daten zu evaluieren.